はじめに
タイトルのままだが、PubMedから検索結果をダウンロードするCLIツールを作ってみた。名称は「ppget」でPythonでPubMedのデータをGetするという意味とした。
例えばターミナルで
ppget "hip spine syndrome sagittal alignment"のように検索語句を入力すると、検索結果のcsvもしくはjsonがダウンロードされる。
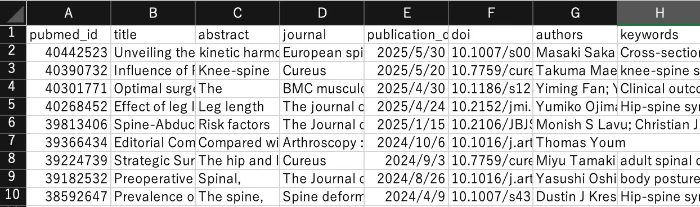
詳細な使い方はREADMEを参照だが、とにかくシンプルかつ簡単に使えるということを重視した。想定用途はシステマティックレビュー、メタアナリシス、テキスト分析などの入口の部分だ。
なぜツールを作ったか
PubMedのサイトからは検索結果を出力することができる。これは簡単に行うことができるのだが、
- テキストファイルかCSVでしか出力できない
- Abstractなどはテキストの羅列としてしか出力できない(構造化データとして出力できない)
といった使いにくさがある。
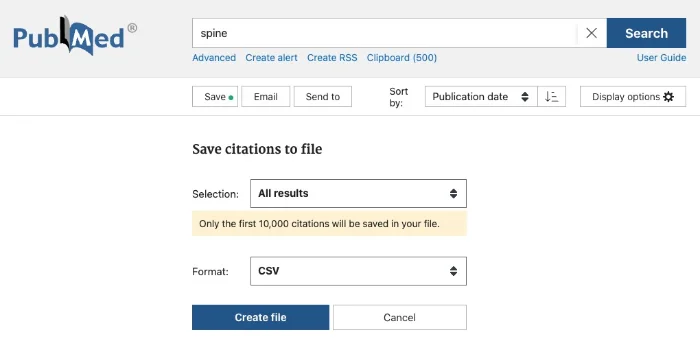
↑PubMedのサイトから検索結果を出力する時
ではデータサイエンティストたちはどうしているかというと、PubMed APIを使っている。PubMed APIを使うのも面倒な部分がある。
- PubMed APIの公式サイトの説明が少ない
- XMLという形式の複雑な階層から必要な情報を切り出さないといけない
- Pythonの環境を準備し、コードを書かないといけない
このうちXMLから必要な情報を切り出す作業に関しては、簡便化するライブラリ(APIラッパー)を配布している人もいる。検索すれば何種類かヒットする。その使い方を覚えれば、それなりに手順を省略化できる。ただ、ちょっとデータ見てみたい時に毎回Pythonのコード書くのはあまりにも面倒だ。そこで、CLIツールを作ることにした。
詳細な話
APIラッパーのうちPyMedが最も簡単そうに見えたが、すでに2020年に開発は終了していた。PyMedからフォークしたリポジトリの1つが継続してバグ対応などをしているようだったので、そのライブラリ(pymed-paperscraper)を利用した。別のデータ解析用のライブラリの依存関係になっているようだった。pymed-paperscraperでXMLをパースして、念の為XMLのフォールバックを組むことにした。CLI化して即座に使えるようにPyPIに登録した。要はAPIラッパーをベースにCLIのオプションを作るだけで良いので、簡単にできそうなやり方だった。現状使う当てはないもののすぐ作れそうだから作ったというのが正直な所だ。
オプション
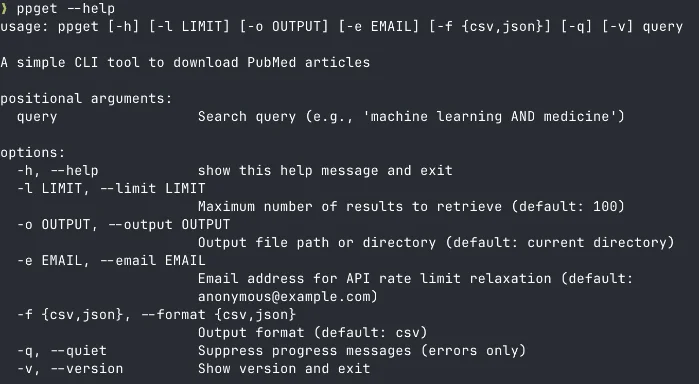
↑ppget —helpで出てくるオプション画面
主なオプションは以下の通り:
-l, --limit:取得件数の指定(デフォルト100件)-f, --format:出力形式の指定(csvまたはjson)-o, --output:出力先のファイルまたはディレクトリ-e, --email:メールアドレス(API制限緩和)
検索クエリにはPubMedの検索構文がそのまま使える(AND、OR、MeSH用語、年指定など)。
おわりに
ppgetを使えば、PubMedからのデータ取得が一行のコマンドで完結する。uvがインストールされている場合はuvx ppget "検索語句"でインストール不要ですぐ使えるのが便利だ。
正直なところ、現時点で明確な使用用途があって作ったわけではない。「思いついてかつ簡単に作れそうだから作った」というのが実情だ。ただ、こういった小さなツールの積み重ね、OSS精神ともいうべきものが研究の世界でもあってほしいと思う。